检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
近日我院李鸿彬/孟状研究团队在《Horticultural Plant Journal》(中科院1区TOP)发表题为“Repetitive sequence landscapes provide insights into the proliferation and reduction of pericentromeric/centromeric repeats in the genus Glycyrrhiza”(重复序列景观图谱揭示甘草属近着丝粒/着丝粒区域的进化动态)的原创性研究论文。课题组已于2024年9月在植物学国际权威期刊《The Plant Journal》(中科院1区TOP)发表关于甘草属种间染色体进化关系和物种起源研究,此次研究成果是甘草属植物染色体进化生物学相关研究又一重要新进展。

重复序列曾一度被称为“垃圾DNA”,然而随着不断的研究发现,重复序列在生物进化、物种形成及基因表达调控中扮演着至关重要的角色。甘草作为最常用的中草药之一,具有极高的药用价值,素有“国老”之称。甘草基因组中重复序列的占比高达61.7%。然而,目前对甘草属植物重复序列结构组成、进化动态及功能意义知之甚少,这严重阻碍了甘草属重复序列演化及功能研究。针对这一问题,该课题组通过甘草低深度测序组装聚类结合荧光原位杂交技术建立了一套甘草重复序列可视化工作体系(图1),并建立了7种甘草主要重复序列的景观图谱。
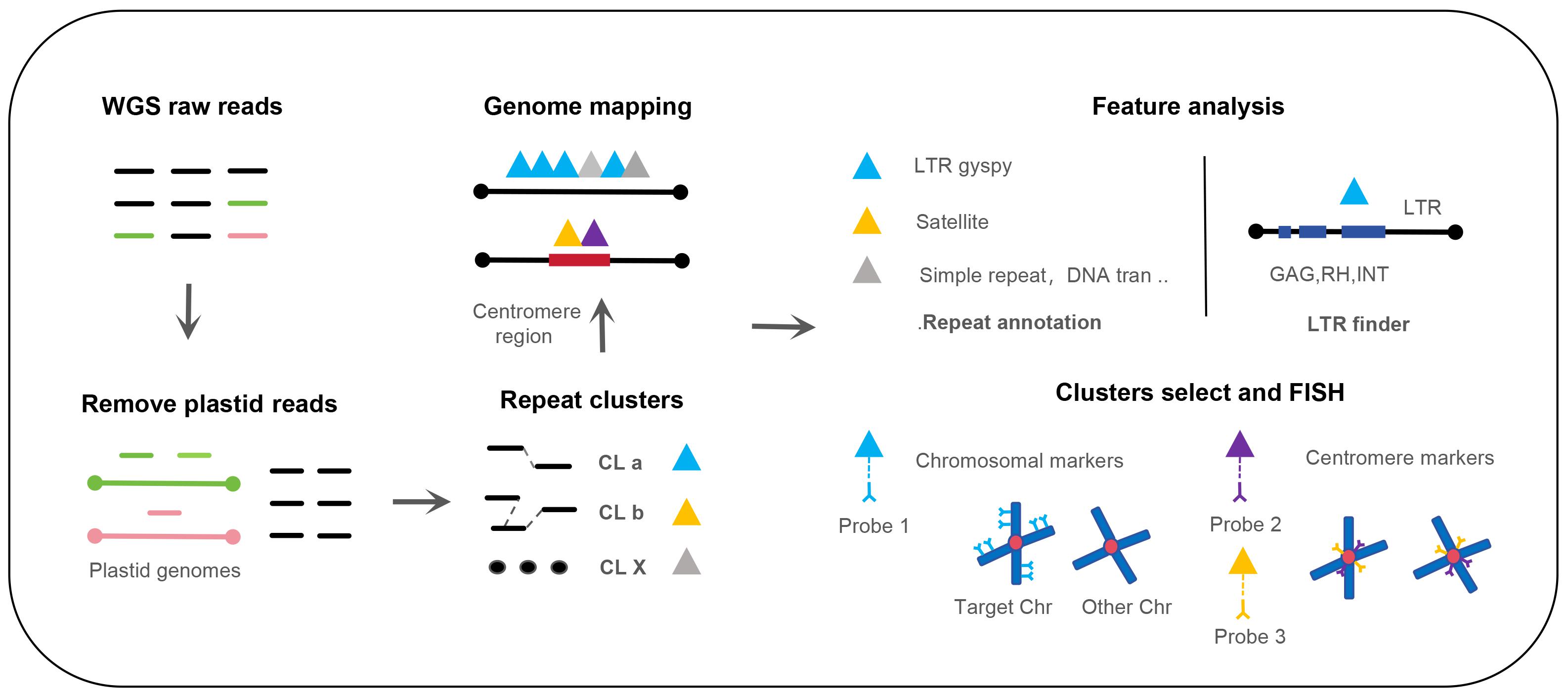
图1. 重复序列可视化工作流程
景观图谱显示多个重复序列实际染色体物理定位与基因组中预测位置存在差异(图2),表明复杂重复序列在当前甘草基因组的精准组装仍然面临巨大挑战。此外,研究还着重分析了6个近着丝粒/着丝粒重复序列在7个甘草种间的进化差异和轨迹,发现年轻的Gly252反转录转座子总是偏好插入到与其末端高度相似的Gly129卫星重复区域,并且在甘草物种独立分化后仍保持协同进化模式(图3)。本研究深入剖析了甘草属种间重复序列的结构组成和进化差异,为未来高质量甘草属植物基因组组装和着丝粒演化相关研究提供了重要参考依据。
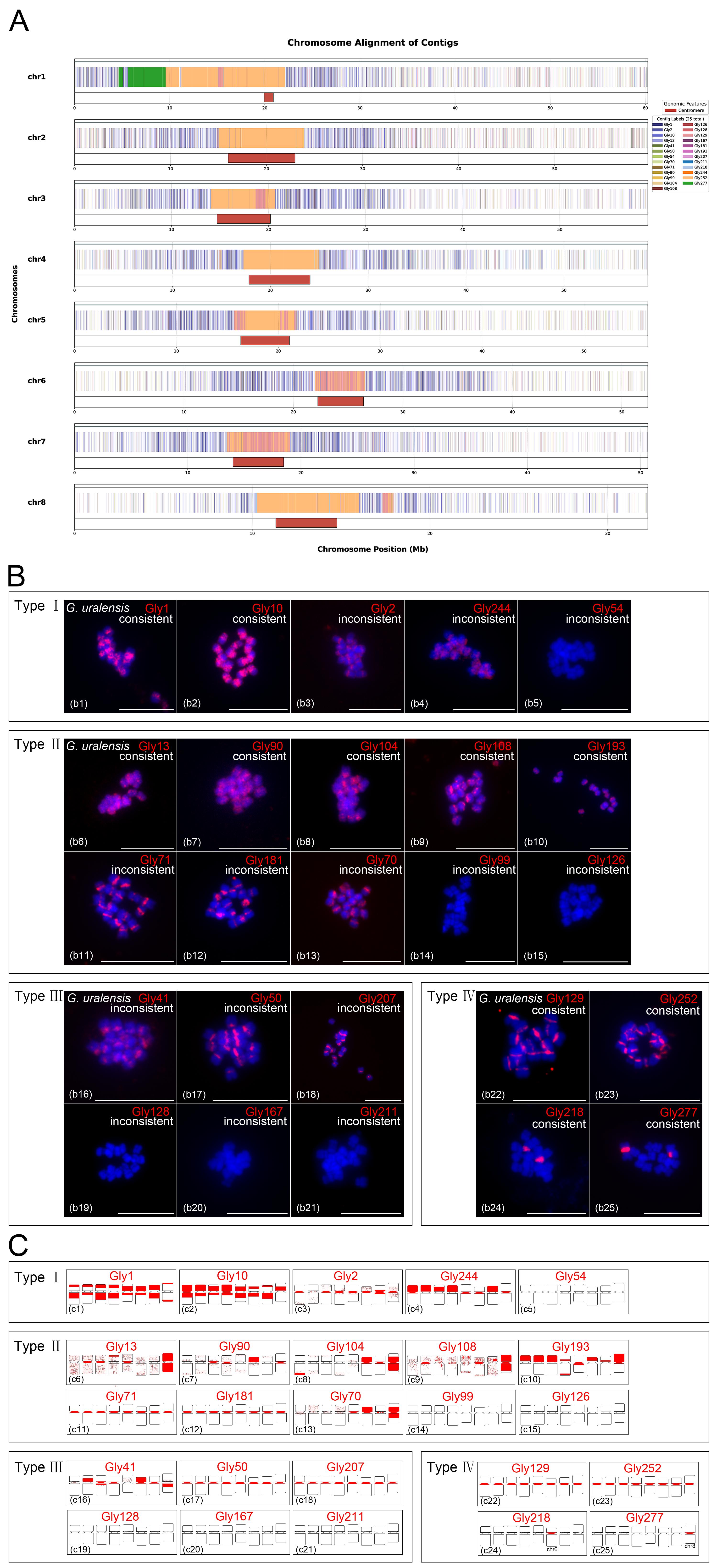
图2. 甘草25个重复序列景观图谱
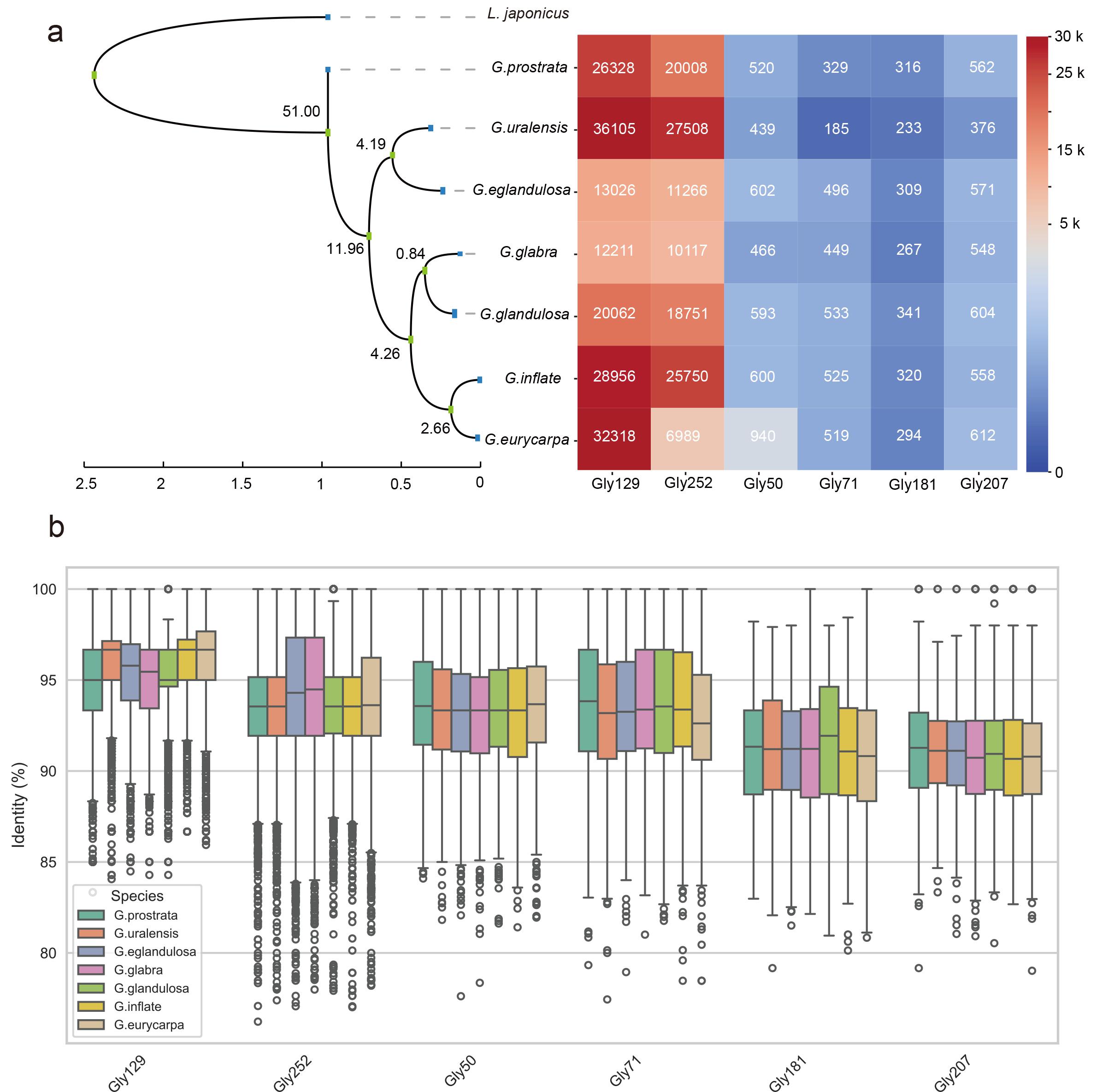
图3. 甘草属植物近着丝粒/着丝粒区域重复序列拷贝数及相似性的动态变化
石河子大学为该论文第一完成单位,石河子大学生命科学学院孟状副教授为论文通讯作者,李鸿彬教授和安徽师范大学生命科学学院左胜副教授为论文共同通讯作者,石河子大学生命科学学院博士生郑仟为论文第一作者,硕士生王伟为论文共同第一作者。该工作得到了兵团自然基金项目(2024DA021)、石河子大学青年拔尖人才项目(BJZK202403)、石河子大学高层次人才科研启动资金项目(RCZK202362)、新疆维吾尔自治区“天池英才”青年博士人才项目(CZ001604)、研究生科研创新项目(BTYJXM-2024-K31)、兵团自然基金项目(2023AB052, 2018AB012, 2020AA005)等的资助。
(通讯员:黄刚 编审:黄刚 胡圣伟 孙辉 终审:胡建松)